
No se trata ya de un solo tronco ancestral del que surgieron todas las ramas de los seres vivos, sino que las principales categorías de organismos estaban ya presentes en el comienzo de los tiempos. Esto es precisamente lo que predice el Diseño inteligente.
![Foto de [link]Johann Siemens[/link], Unsplash.](https://cms.protestantedigital.com/upload/imagenes/6286b97877377_1.jpg) Foto de [link]Johann Siemens[/link], Unsplash.
Foto de [link]Johann Siemens[/link], Unsplash.
No cabe duda de que actualmente el principal apoyo que tiene la teoría de la evolución proviene de la biología molecular y la genética. Tal como se señaló anteriormente, el hecho de que ciertos trozos de material hereditario se encuentren, por ejemplo, en simios y humanos se interpreta como evidencia de que ambos descienden de un antepasado común. El desarrollo de estas disciplinas se produjo sobre todo a partir de la década de los 50 del pasado siglo. En el año 1953, el biólogo estadounidense James Watson y el físico británico Francis Crick descubrieron la singular estructura helicoidal del ácido desoxirribonucleico (ADN) que conforma los cromosomas del núcleo de las células. El hecho más misterioso de la biología moderna es que esta macromolécula contiene la información biológica necesaria para la existencia de cada especie animal o vegetal. Nadie sabe cómo semejante volumen de información sofisticada pudo originarse por primera vez porque entender cómo se transmite dicha información a las proteínas no explica, ni mucho menos, cómo surgió al principio.
[ads_google]div-gpt-ad-1623832500134-0[/ads_google]
Los ladrillos de la información genética son las cuatro famosas bases nitrogenadas del ADN (adenina, timina, citosina y guanina) que forman los nucleótidos que lo componen. Esta información se transmite al ARN (que en vez de timina tiene uracilo) y a partir de éste se determina la secuencia de aminoácidos de las proteínas, que son las encargas de realizar todas las funciones vitales de los organismos. De manera que si se quiere estudiar la información biológica de las especies hay dos formas de hacerlo: examinando directamente la molécula de ADN o bien estudiando la cadena de aminoácidos de las proteínas a que éste da lugar.
Esta comparación de la secuencia de nucleótidos en el ADN entre especies biológicas, o de los aminoácidos de sus proteínas, se interpreta desde el evolucionismo como indicativa de las divergencias o parecidos filogenéticos entre los diferentes organismos. Se supone que si más se parecen las moléculas de dos especies diferentes es porque éstas tienen mayor parentesco evolutivo. En base a este criterio, se han elaborado árboles filogenéticos que supuestamente reflejarían las relaciones evolutivas entre los seres vivos.
Por ejemplo, el citocromo c es una proteína respiratoria pequeña de sólo 104 aminoácidos que está presente en todos los seres vivos y cuya misión es transportar electrones en las mitocondrias de las células. Pues bien, en base al número de aminoácidos diferentes que presenta esta proteína en las distintas especies biológicas se elaboró un árbol genealógico (o filogenético) que supuestamente indicaría el parentesco evolutivo existente entre dichas especies. Según este árbol, los monos estarían más próximos a los humanos que los caballos. Lógicamente esto ya se suponía desde la anatomía comparada, la paleontología y la simple lógica transformista. No obstante, también aparecían datos contradictorios con los árboles clásicos, como que las palomas y los perros estuvieran a la misma distancia de los humanos en cuanto al número de cambios sufridos en los aminoácidos del citocromo c. ¿Cómo podían estar a la misma distancia filogenética del hombre un mamífero y un ave? Lo mismo ocurría con los canguros, mamíferos marsupiales que salían más cercanos al ser humano que los perros, que son mamíferos placentarios como nosotros.
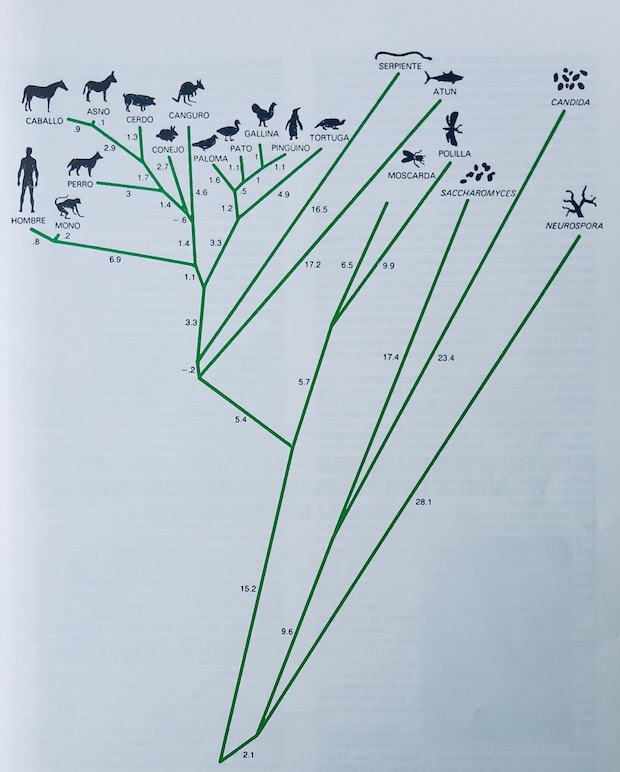
[photo_footer]Primer árbol filogenético generado con computador y basado en las diferencias en la secuencia de aminoácidos del citocromo c de una veintena de organismos distintos. Fue elaborado en 1967 por Walter M. Fitch de la Universidad de Wisconsin y Emanuel Margoliash de la Northwestern University.[1] [/photo_footer]
Cuando se pasa de las comparaciones morfológicas (aspecto físico de los animales) a los parecidos entre secuencias moleculares (sean éstas de genes, pseudogenes, nucleótidos o aminoácidos de las proteínas) surgen también muchas dificultades. Lógicamente el darwinismo se fija en aquellas que parecen corroborar la idea del ancestro común y descarta todas las que contradicen tal supuesto, pero éstas últimas son tan abundantes que han colocado en entredicho todo el árbol de la vida propuesto por Darwin. En vez de un solo tronco con muchas ramas, la genómica ha dibujado un zarzal de múltiples raíces y ramas conectadas entre sí a varios niveles.
Todos los seres vivos, desde las bacterias al hombre, poseemos ADN constituido por las famosas cuatro bases nitrogenadas (A, T, C y G) y el orden de éstas determina la secuencia de los aminoácidos en las proteínas. Cuando las células se reproducen, o dividen, se copian perfectamente estas secuencias de bases nitrogenadas. Sin embargo, a veces ocurren accidentes o mutaciones que generan copias ligeramente diferentes a las de las células progenitoras. Esto significa que los organismos podemos tener moléculas de ADN e incluso proteínas que pueden diferir algo de las de nuestros padres, abuelos o tatarabuelos. En 1965 se sugirió desde el evolucionismo que tales diferencias genéticas podrían ser útiles para determinar las relaciones evolutivas entre especies diferentes.[2] Aquellas cuyas proteínas o ADN difirieran poco estarían supuestamente más estrechamente relacionadas, en términos evolutivos, que quienes lo hicieran en un mayor número de subunidades.
[ads_google]div-gpt-ad-1623832402041-0[/ads_google]
Después de comparar durante años los resultados obtenidos con distintos árboles genealógicos para proteínas como los citocromos, globinas, hemoglobinas, genes, cromosomas, etc., el famoso paleontólogo evolucionista inglés Colin Patterson confesó que: “una computadora (convenientemente programada) puede dibujar una genealogía a partir de secuencias proteínicas de origen desconocido. Es importante no caer en el error de pensar que estos árboles de secuencias de proteínas, por cuanto están calculados mediante computadoras y contienen cifras concretas, representan el curso actual de la evolución. No es así. El árbol producido por computadora no es necesariamente más cierto o factible que uno diseñado por un anatomista comparativo con su estudio de la estructura de una gran variedad de especies. Los dos árboles pueden constituir un valioso test uno para el otro, pero sin embargo pueden no ser mejores que la persona que observó las especies o la que escribió el programa del computador. La calculadora puede dibujar, a partir de una misma secuencia de datos, una gran variedad de árboles, según el tipo de asunciones que haya hecho el programador.”[3]
Al asumir que las mutaciones se han venido acumulado de forma constante a lo largo del tiempo, entonces el número de diferencias entre los organismos podría servir como una especie de "reloj molecular" que indicara incluso cuántos años habrían transcurrido desde que su ADN o proteínas eran idénticos. Es decir, cuánto tiempo hace que compartieron un antepasado común. El problema es que la comparación de secuencias de ADN de los distintos organismos vivos que existen hoy es una tarea compleja ya que contienen miles de subunidades (bases nitrogenadas) y alinearlas correctamente es difícil. Según como se alineen, pueden dar resultados muy diferentes.
Por ejemplo, cuando se intentó datar la famosa explosión biológica del Cámbrico, es decir, la aparición súbita de todas las clases de seres vivos según atestiguan los fósiles, por medio de este reloj molecular, los diversos investigadores llegaron a resultados muy dispares. Unos situaban el antepasado común de todos los seres vivos entre los 900 y 1000 millones de años de antigüedad.[4] Otras comparaciones moleculares recortaban esta fecha a los 670 millones de años.[5] Mientras que algunos científicos retrasaban a 1200 millones de años el tiempo en que supuestamente vivió el ancestro común.[6] ¿Cómo es posible que los relojes moleculares mostraran un retraso de 530 millones de años, entre los resultados máximos y mínimos? ¿No indicaba esta amplia gama de posibilidades que tales relojes no son de fiar?
Desde la década de los 70 del pasado siglo, los biólogos evolucionistas han estado esperando que las comparaciones de secuencias de ADN entre las distintas especies superaran estas dificultades y permitieran construir un árbol de la vida universal que corroborara de alguna manera sus predicciones teóricas. Sin embargo, la mayoría de los descubrimientos han venido frustrando esta esperanza. Cuanto más secuencias se comparan, tanto de ADN como de ARNr (ácido ribonucleico ribosómico) o de proteínas, las filogenias resultantes más se contradicen entre sí y con el supuesto árbol evolutivo de la vida.[7] Las diversas moléculas rastreadas conducen a árboles filogenéticos muy diferentes.
Un artículo científico publicado en 1996, que analizaba 88 secuencias distintas de proteínas, situaba a los conejos más cerca de los primates que de los demás roedores.[8] Otro estudio de trece genes en 19 especies animales colocaba a los erizos de mar (invertebrados) entre los cordados (que incluyen a los vertebrados). Y, en fin, de otro basado en doce proteínas distintas se desprendía que las vacas están más cerca de las ballenas que de los caballos.[9] Tales inconsistencias han sumido la filogenia molecular en una grave crisis. Se ha sugerido que quizás el hipotético antepasado común universal no era un organismo vivo sino un proceso metabólico que podía intercambiar material genético o que las discrepancias en las filogenias moleculares pueden deberse a la transferencia lateral de genes entre especies distintas. Pero lo que está claro es que el darwinismo necesita nuevas hipótesis, que aún no se vislumbran, que sean capaces de solucionar satisfactoriamente tales problemas. Hoy por hoy, la evidencia fósil y molecular demuestra que el ramificado árbol de la evolución propuesto por Darwin es absolutamente inconsistente.
La explosión de vida del cámbrico confirma que los filos o categorías más altas de la clasificación de los seres vivos aparecieron al principio y no después de cientos de millones de años de evolución. Esto arranca el árbol de la evolución y lo vuelve del revés. No se trata ya de un solo tronco ancestral del que surgieron todas las ramas de los seres vivos sino que las principales categorías de organismos estaban ya presentes en el comienzo de los tiempos y han podido cambiar relativamente. Esto es precisamente lo que predice el Diseño inteligente. Es decir, que la complejidad de los seres vivos, aunque éstos hayan podido variar en el tiempo, existe desde los orígenes.
Notas
[1] Fitch, W. M. & Margoliash, E. 1967, Construction of Phylogenetic Trees, Science, 20 Jan 1967: Vol. 155, Issue 3760, pp. 279-284.
[2] Zuckerkandl, E. & Pauling, L., 1965, Molecules as Documents of Evolutionary History, Journal of Theoretical Biology, 8, pp. 357-366.
[3] Patterson, C. 1985, Evolución: La teoría de Darwin hoy, Fontalba, Barcelona, p. 137-139.
[4] Runnegar, B., 1982, A molecular clock date for the origin of the animal phyla, Lethaia, 15, pp. 199-205.
[5] Doolittle, R. F., et al., 1996, Determining Divergence Times of the Major Kingdoms of Living Organisms with a Protein Clock, Science, 271, pp. 470-477.
[6] Wray, G. A., et al., 1996, Molecular Evidence for Deep Precambrian Divergences Amoung Metazoan Phyla, Science, 274, pp. 568-573.
[7] Lake, J. A. et al., 1999, Mix and Match in the Tree of Life, Science, 283, pp. 2027-2028; Phillippe, H. & Forterre, P., 1999, The Rooting of the Universal Tree of Life Is Not Reliable, Journal of Molecular Evolution, 49, pp. 509-523.
[8] Graur, D. et al., 1996, Phylogenetic position of the order Lagomorpha (rabbits, hares and allies), Nature, 379, pp. 333-335.
[9] Cao, Y. et al., 1998, Conflict Amoung Individual Mitochondrial Proteins in Resolving the Phylogeny of Eutherian Orders, Journal of Molecular Evolution, 47, pp. 307-322.
 La década en resumen: teología, con José Hutter
La década en resumen: teología, con José Hutter
La conmemoración de la Reforma, las tensiones en torno a la interpretación bíblica de la sexualidad o el crecimiento de las iglesias en Asia o África son algunos de los temas de la década que analizamos.
 Intervalos: Disfruten de la luz
Intervalos: Disfruten de la luz
Estudiamos el fenómeno de la luz partiendo de varios detalles del milagro de la vista en Marcos 8:24, en el que Jesús nos ayuda a comprender nuestra necesidad de ver la realidad claramente.
 2020, año del Brexit
2020, año del Brexit
Causas del triunfo de Boris Johnson y del Brexit; y sus consecuencias para la Unión Europea y la agenda globalista. Una entrevista a César Vidal.
 7 Días 1x08: Irak, aborto el LatAm y el evangelio en el trabajo
7 Días 1x08: Irak, aborto el LatAm y el evangelio en el trabajo
Analizamos las noticias más relevantes de la semana.
 Min19: Infancia, familia e iglesias
Min19: Infancia, familia e iglesias
Algunas imágenes del primer congreso protestante sobre ministerios con la infancia y la familia, celebrado en Madrid.
 X Encuentro de Literatura Cristiana
X Encuentro de Literatura Cristiana
Algunas fotos de la entrega del Premio Jorge Borrow 2019 y de este encuentro de referencia, celebrado el sábado en la Facultad de Filología y en el Ayuntamiento de Salamanca. Fotos de MGala.
 Idea2019, en fotos
Idea2019, en fotos
Instantáneas del fin de semana de la Alianza Evangélica Española en Murcia, donde se desarrolló el programa con el lema ‘El poder transformador de lo pequeño’.
 Héroes: un padre extraordinario
Héroes: un padre extraordinario
José era alguien de una gran lealtad, la cual demostró con su actitud y acciones.
 Programa especial de Navidad en TVE
Programa especial de Navidad en TVE
Celebración de Navidad evangélica, desde la Iglesia Evangélica Bautista Buen Pastor, en Madrid.
 Primer Congreso sobre infancia y familia, primera ponencia
Primer Congreso sobre infancia y familia, primera ponencia
Madrid acoge el min19, donde ministerios evangélicos de toda España conversan sobre los desafíos de la infancia en el mundo actual.

Las opiniones vertidas por nuestros colaboradores se realizan a nivel personal, pudiendo coincidir o no con la postura de la dirección de Protestante Digital.
Si quieres comentar o